우선 데이터 분석에 앞서, Chromosome conformation capture이 무엇이고 왜 중요한지에 대해 이야기하고자 한다.
지난 수십여 년간 엄청난 발전을 거듭하고 있는 sequencing technology들로 인해 우리는 유전자 서열에 대한 정보를 높은 해상도로 이해하기 시작하였다. 그로 인해, 유전자 염기서열의 변이들이 미치는 영향들에 대해 많은 정보들 얻게 되었지만 더불어 염기 서열의 특성이나 후성유전학으로 설명하기 어려운 현상들 또한 발견하게 되었다.
특히 인간 전체 염기서열의 98%를 차지하는 non-coding region에서 일어나는 변이가 어떠한 원인으로 표현형에 영향을 미치는지에 대해 많은 노력을 기울이기 시작하였다. 그러면서 promoter, enhancer, motif 등과 같이 유전자의 발현을 조절하는 인자들에 대한 이해와 더불어, genome에 얼마나 조밀 또는 느슨하게 histone 단백질에 감싸져 있는지, genomic elements들 사이의 물리적 거리에 따라 달라지는 유전자들의 발현에 대해 관심을 가지게 되었다. 이와 같은 접근은 genome을 ATGC 염기서열을 단순히 배열하는 것이 아니라 더 높은 차원의 구조적 이해를 필요로 하게 되었다.
이로 인해, 3C (chromosome conformation capture)에서 부터 4C (chromosome conformation capture-on-chip/circular chromosome conformation capture) 그리고 5C (chromosome conformation capture carbon copy)와 같이 기술들이 차례로 개발되었다. 3C의 경우에는 유전체의 특정 부분의 interaction만을 볼 수 있지만, 4C에서는 한 부분에 대응하는 여러 interaction을 그리고 5C에서는 유전체의 여러 부분의 interaction을 동시에 볼 수 있게 되었다. 하지만 여전히 유전체 전반에 걸친 interaction을 보기에는 무리가 있었고 이를 2009년 Aiden이 NGS를 이용한 Hi-C 분석법을 통해 3D genomics 시대가 활짝 열리게 되었다.

library read depth에 따라 Hi-C analysis를 통해 chromatin의 특징을 3 Mb의 은 scale에서 부터 100Mb 이상의 whole genome scale까지 접근하는 것이 가능하게 되었고, scale에 따라, Chromosome territories, Hubs와 compartment, TAD와 loop domain와 같은 정보를 얻을 수 있게 되었다.

Genome-wide scale에서 활발히 활용되는 것은 whole genome assembly라 볼 수 있다. 지금까지 오랜 시간 축적되어온 short read 들과, 최근 엄청난 진보를 이루고 있는 Pacbio 또는 Nanopore long read sequencing 기술들에 힘입어, 전체 유전체 서열을 읽고, trait 들을 찾는 EARTHBIOGENOME, ZOOnomia 와 같은 거대 project들이 수행되고 있는데, 염기서열을 읽고 이를 조립하는 과정에서 Hi-C read가 제공하는 intrachromosomal interaction이 interchromosomal interaction 정보들을 이용하여 더 높은 정확도로 염색체상의 위치와 방향성을 얻을 수 있다.
이외에도 Hi-C 분석을 통해 이해할 수 있는 것은 기존의 short read sequencing으로는 detection에 제약이 있는 structural variant (SV, e.x., deletion, duplication, inversion)들을 TAD 영역의 변화를 통해 detection 하게 되었을 때, 해당 영역의 유전자 발현 등을 추가로 분석함으로써 SV로 인해 일어나는 표현형 변화와 그 원인관계에 대해 더 깊이 이해할 수 있게 된다.
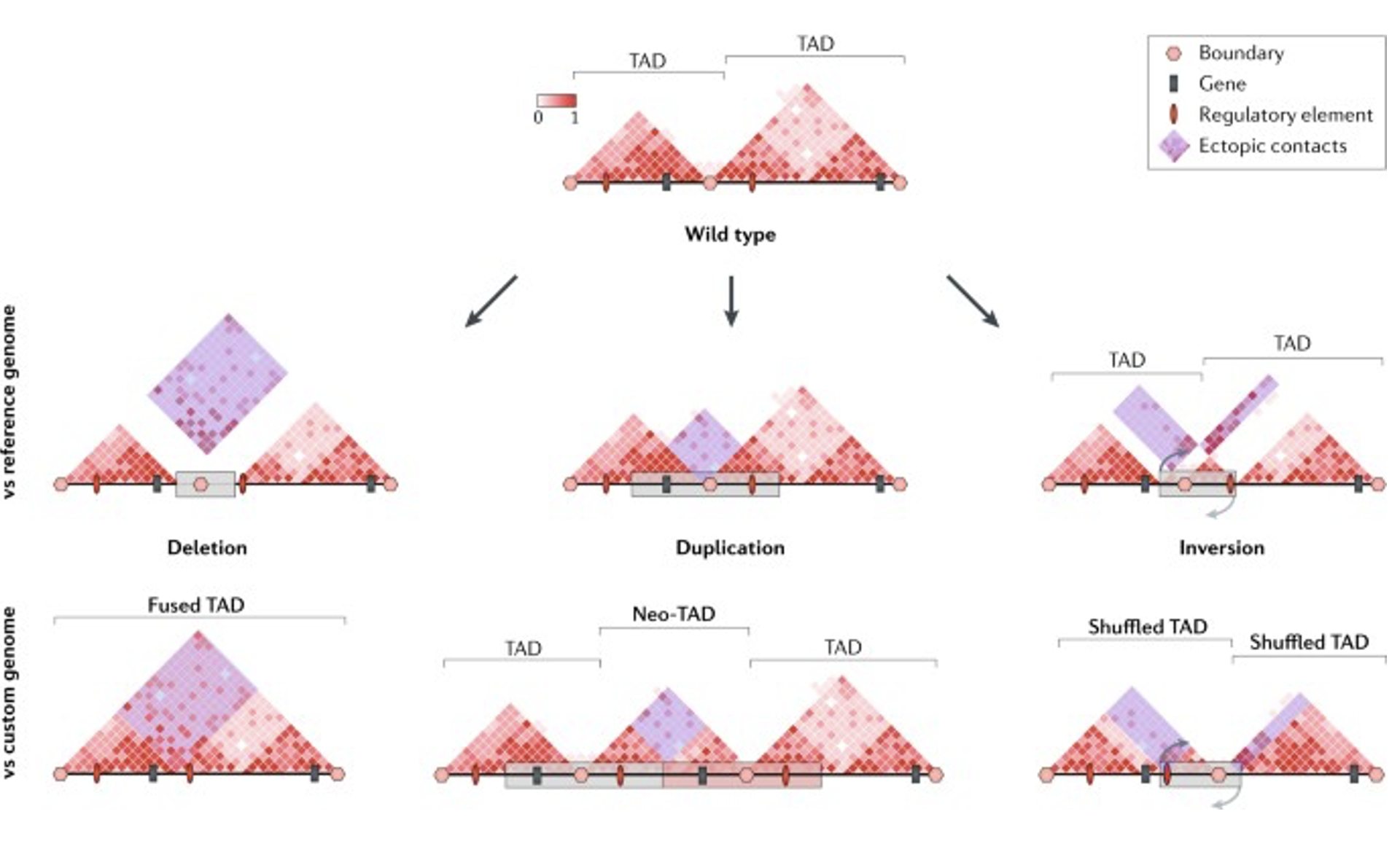
TAD: Topological association domain TAD는 self-interacting genomic region으로 그 말인 즉, 같은 TAD 안에 있는 DNA sequence는 다른 TAD안에 있는 DNA sequence 보다 물리적으로 더 많은 interaction을 한다. 이러한 TAD의 양쪽 끝단에는 CTCF binding factor와 cohesion이 존재하는데, 이는 DNA를 잡고 압출하는 형식으로 loop를 형성하여, DNA가 물리적으로 가까워질 수 있도록 한다. 따라서, 이 TAD 내부에 있는 motif들은 멀리 떨어져 있어도 물리적으로 가까이 위치할 수 있기 때문에 transcription factor들과 함께 작용 시에 유전자의 발현에 직접적으로 관여할 수 있다. 아래의 Figure는 지금까지 밝혀진 SV로 인해 일어나는 TAD의 변화가 유전자의 발현에 영향을 미치고 phenotype에 영향을 준 임상 결과들이다.

과학 기술의 발전으로 이전에는 감히 도달하지 못했던 영역까지 볼 수 있는 window를 제공하는 동시에
새로운 가설과 상상력을 자극하며 더 많은 것들에 대해 이해 하도록 촉진하고 있다.
앞으로 우리가 얼마나 더 많은 흥미로운 사실들을 발견하게 될까?
'실험실 > 실험(Experiments)' 카테고리의 다른 글
| Higlass (0) | 2023.08.18 |
|---|---|
| Hi-C experiments_library prep (0) | 2023.07.27 |
| Bioinformatic (0) | 2021.08.18 |
| 선배, ligation이 안되는데요? (0) | 2020.11.23 |
| PCR-LFA (0) | 2020.09.15 |



